No se ha encontrado investigadores para este término.
En nuestro laboratorio tenemos la convicción de que muchos aspectos de la salud humana no están predeterminados genéticamente. Planteamos que la aparición de enfermedades asociadas al envejecimiento es en parte consecuencia del deterioro de la memoria epigenética celular, lo que finalmente conduce a cambios en la identidad celular y al inicio de patologías asociadas al envejecimiento, incluido el cáncer (Figura 1). Nuestro objetivo es contribuir a establecer los fundamentos moleculares de la memoria epigenética, identificar cuáles son las vías epigenéticas clave que impulsan el envejecimiento y la progresión del cáncer, y al diseño de nuevas terapias que reviertan las enfermedades asociadas al envejecimiento.
Somos un grupo multidisciplinar adscrito al Departamento de Bioquímica y Biología Molecular II de la Universidad de Granada.
Más información: http://www.landeiralab.ugr.es
Actualmente desarrollamos tres líneas de investigación en el laboratorio:
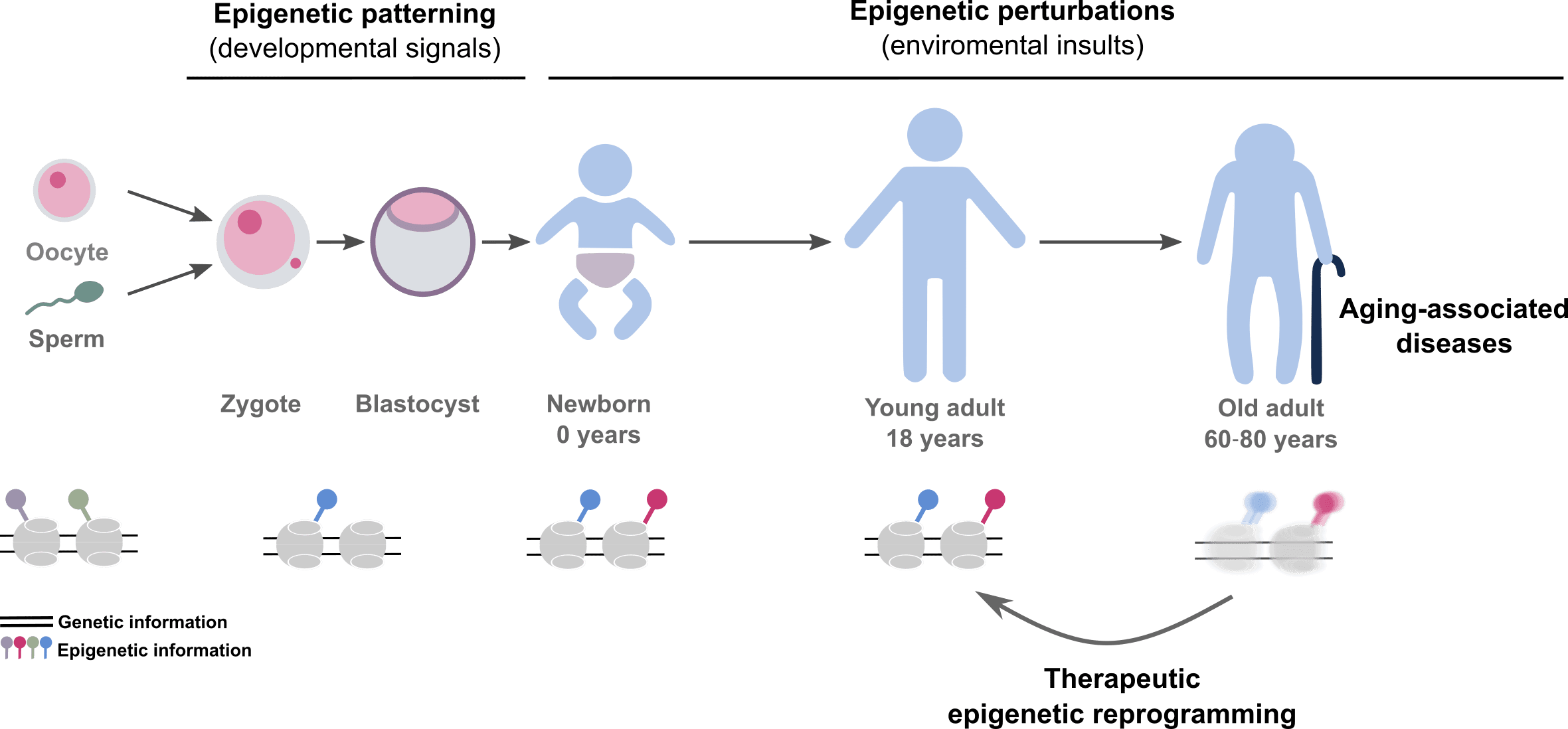
Figura 1. Esquema que resume el interés general del laboratorio en epigenética durante el desarrollo embrionario y en la enfermedad humana.
¿Cuál es la base molecular de la memoria epigenética?
Para tratar enfermedades de base epigenética, primero debemos establecer las rutas epigenéticas clave que facilitan la formación y el funcionamiento de organismos multicelulares complejos como los humanos. Tras la fusión del espermatozoide y el ovocito se forma una única célula llamada cigoto. El cigoto posee un genoma único y, durante el desarrollo del organismo, atraviesa un gran número de rondas de proliferación y diferenciación que generan billones de células genéticamente idénticas, pero funcionalmente distintas, que se autoorganizan para producir los órganos que componen el cuerpo humano adulto. La especialización celular depende de la existencia de diferentes configuraciones epigenéticas que facilitan la expresión de conjuntos de genes específicos de cada tipo celular durante el desarrollo embrionario. Por tanto, los patrones epigenéticos específicos de tipo celular deben “memorizarse” y mantenerse durante toda la vida, porque la señal de diferenciación original que instruyó el cambio de expresión génica en la vida prenatal ya no está presente en la vida adulta. Es importante destacar que distintos estímulos ambientales pueden perturbar la memoria epigenética de las células; por ello, hipotetizamos que muchas enfermedades asociadas al envejecimiento son consecuencia de la perturbación de la configuración epigenética original que se estableció en el genoma durante el desarrollo.
En nuestro laboratorio estamos analizando el mecanismo por el cual los reguladores epigenéticos promueven el mantenimiento de la memoria epigenética. En particular, estudiamos los detalles moleculares de cómo dos sistemas epigenéticos emblemáticos (las proteínas Polycomb y las metiltransferasas de H3K9) permiten la perpetuación de la memoria transcripcional en células madre pluripotentes de ratón. Nuestros hallazgos recientes (1,2) sugieren que la memoria celular se sustenta en un ciclo epigenético en el que actividades reversibles llevadas a cabo por reguladores epigenéticos, coordinadas con las transiciones del ciclo celular, crean un sistema que permite tanto el mantenimiento de la identidad celular como la diferenciación celular durante el desarrollo (3) (Figura 2).
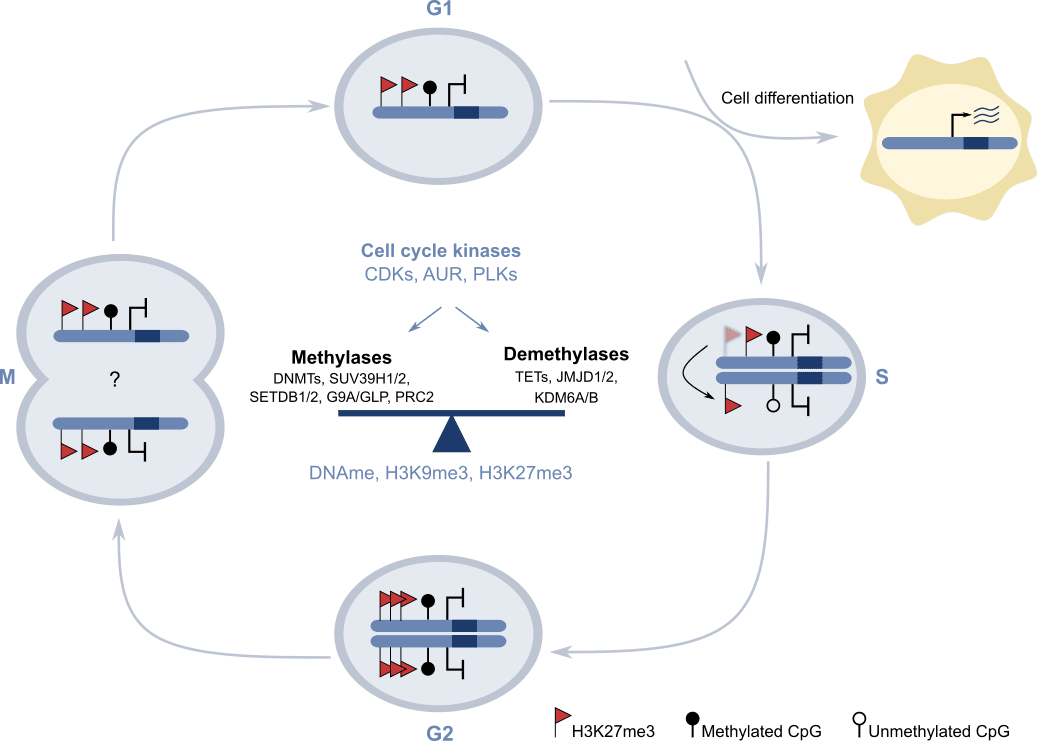
Figura 2. Modelo que propone la base molecular de la memoria epigenética: un “ciclo epigenético” en el que los reguladores epigenéticos se coordinan con las quinasas celulares durante las transiciones del ciclo celular para permitir tanto la autorrenovación como la diferenciación en células madre. Adaptado de Espinosa-Martínez et al., Science Advances 2020.
¿Es la pérdida de información epigenética la razón por la que envejecemos? ¿Podemos ralentizar el envejecimiento con fármacos que estabilicen nuestro epigenoma?
Descifrar las bases moleculares del envejecimiento humano es un reto apasionante de la biología fundamental con grandes implicaciones en salud pública. Descubrimientos recientes sugieren que el envejecimiento podría ser inducido por factores ambientales que comprometen la estabilidad de la información epigenética almacenada en nuestros genomas. Por tanto, apoyamos la idea de que el proceso de envejecimiento está impulsado por la pérdida progresiva de la “información epigenética juvenil” establecida durante el desarrollo, cuya recuperación mediante reprogramación epigenética puede mejorar la función de tejidos dañados y envejecidos al propiciar “la reversión” de la edad (4) (Figura 3).
Utilizando células pluripotentes y ratones mutantes como sistemas modelo, estamos investigando cuáles son los factores epigenéticos que conforman nuestro epigenoma juvenil y que nos protegen del envejecimiento a lo largo de la vida. Por ejemplo, basándonos en un trabajo previo que mostraba que la función de la proteína del reloj molecular BMAL1 durante el desarrollo es crucial para evitar el envejecimiento precoz en ratones adultos (5), hemos descubierto que en células pluripotentes BMAL1 se reutiliza como represor transcripcional de elementos transponibles a través de H3K9me3, independientemente de CLOCK (6), lo que pone de manifiesto que las perturbaciones epigenéticas durante la vida prenatal pueden tener un impacto drástico en la salud y la longevidad de los mamíferos. Prevemos que la caracterización de estos mecanismos ofrecerá una mejor comprensión de las bases moleculares del envejecimiento y permitirá diseñar intervenciones farmacológicas seguras que nos protejan frente a la pérdida de la memoria epigenética que codifica la juventud a lo largo de la vida.
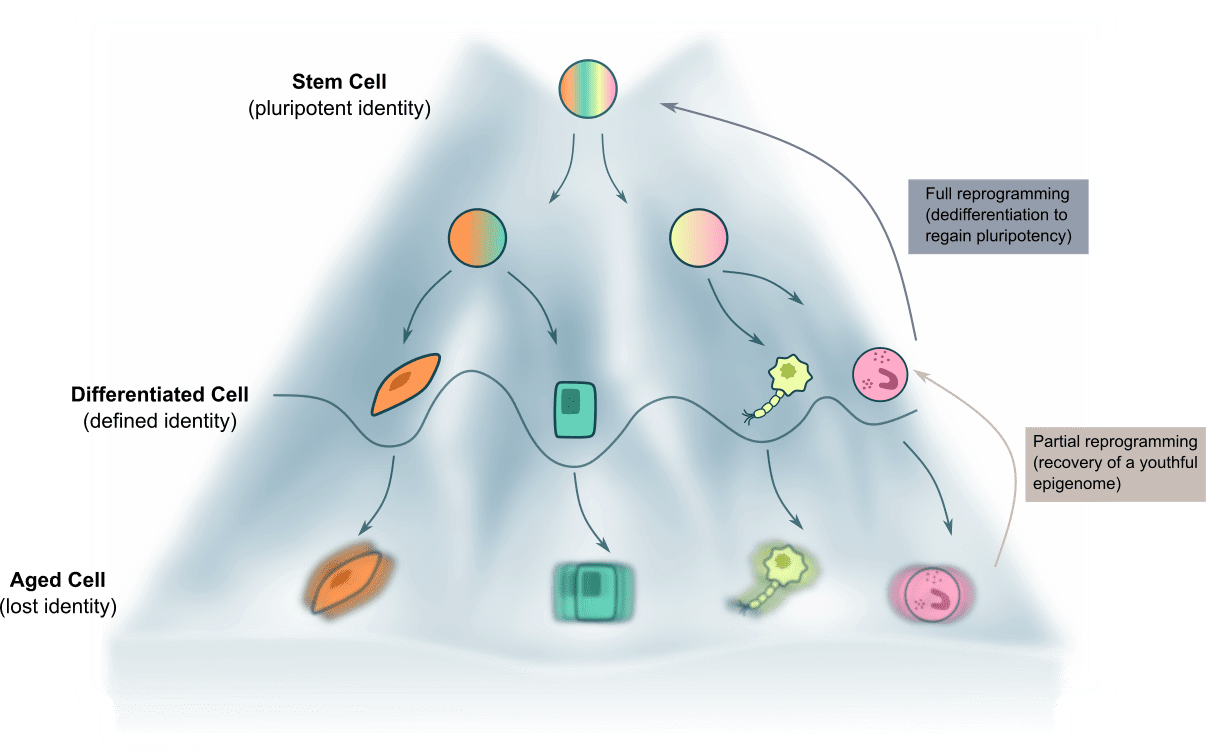
Figura 3. Esquema que propone transiciones en el paisaje epigenético del desarrollo, el envejecimiento y el rejuvenecimiento. Inspirado en la metáfora original de Waddington, los valles representan estados de identidad celular más estables que están separados entre sí por “muros” (el epigenoma) durante la especificación del desarrollo. A medida que envejecemos, esos muros se alteran y la identidad celular se vuelve menos estable. Se indican estrategias de reprogramación que favorecen el restablecimiento del epigenoma. Adaptado de Ryan Lu et al., Nature Aging 2023.
¿Es la inestabilidad epigenética un motor de la progresión tumoral? ¿Podemos curar el cáncer modulando el epigenoma del cáncer?
El cáncer es una de las enfermedades más prevalentes en poblaciones envejecidas. La metástasis causa ~90% de la mortalidad asociada al cáncer y, por tanto, para curarlo es urgente desarrollar nuevas terapias que bloqueen la capacidad de las células tumorales de diseminarse por el organismo. La diseminación metastásica se basa en la selección positiva continua de células cancerosas funcionalmente optimizadas para sobrevivir en los distintos entornos fisiológicos y farmacológicos que aparecen durante la progresión de la enfermedad. En nuestro laboratorio creemos que la heterogeneidad funcional entre células, que impulsa la progresión tumoral, se sustenta no solo en combinaciones diferentes de mutaciones genéticas acumuladas en su genoma, sino también en configuraciones epigenéticas distintas que dictan el comportamiento de cada célula (7).
Utilizamos células tumorales derivadas de pacientes y modelos de xenotrasplante en ratón para investigar cómo la memoria epigenética afecta a la progresión del cáncer, con el objetivo de contribuir al desarrollo de nuevas terapias que frenen la enfermedad utilizando farmacos que modulen la actividad de reguladores epigenéticos. Recientemente hemos demostrado que los reguladores epigenéticos Polycomb aportan plasticidad fenotípica epitelio-mesénquima en células tumorales, facilitando así la diseminación metastásica (8,9) (Figura 4). Estos descubrimientos son importantes para la aplicación eficaz de los inhibidores de Polycomb que están actualmente disponibles para uso clínico en pacientes humanos con cáncer.
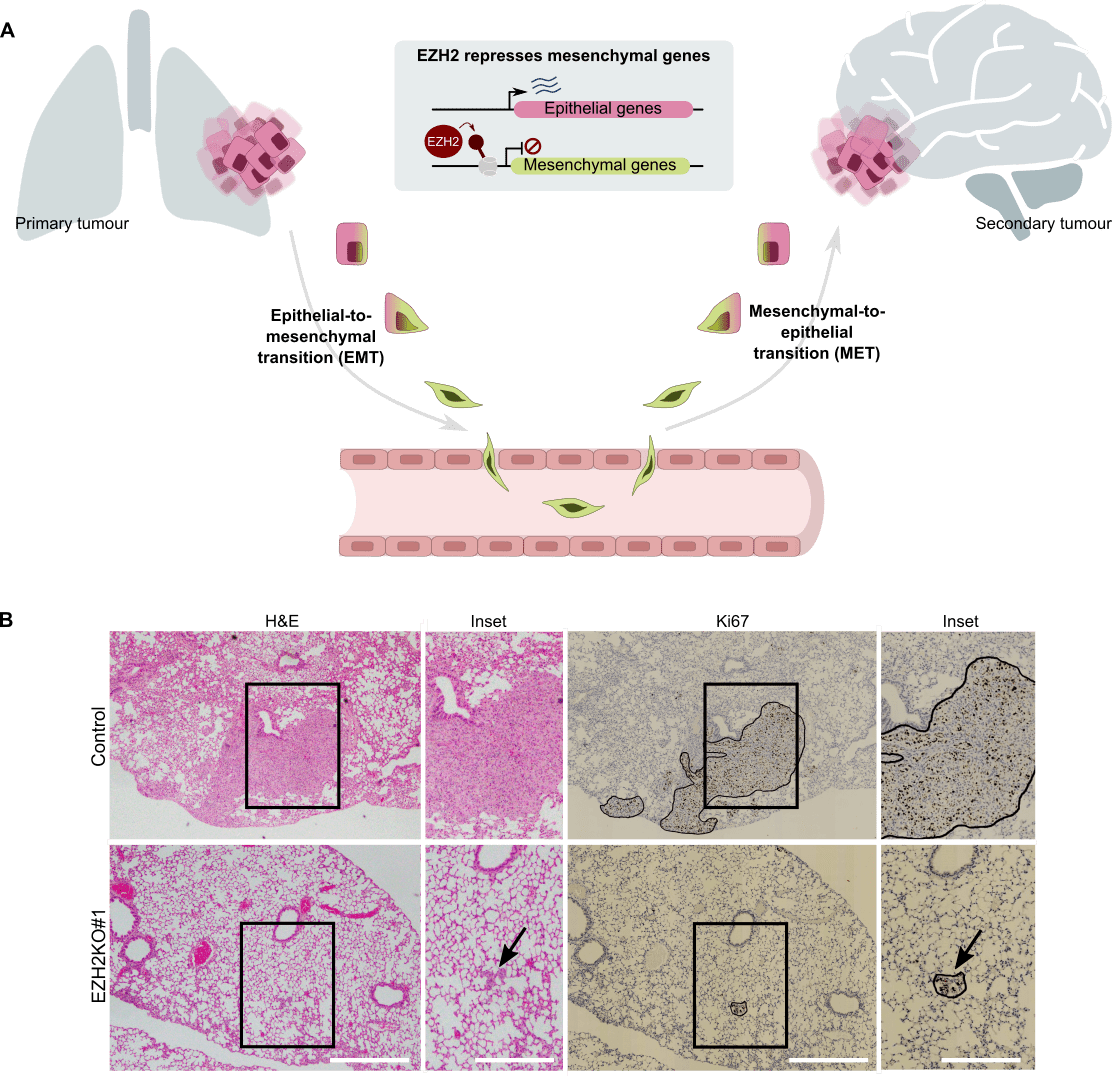
Figura 4. Regulación de la metástasis por el regulador epigenético EZH2. A) Esquema que resume el papel de la proteína Polycomb EZH2 en la regulación de la transición epitelio-mesénquima durante la metástasis de carcinomas. B) Imágenes de inmunohistoquímica de tumores formados en pulmones de ratón que demuestran que las células de cáncer de pulmón requieren EZH2 para una colonización tumoral eficiente. Adaptado de Gallardo et al., Oncogene 2023.
Bibliografia
1. H. G. Asenjo, M. Alcazar-Fabra, M. Espinosa-Martínez, L. Lopez-Onieva, A. Gallardo, E. Dimitrova, A. Feldmann, T. Pachano, J. Martorell-Marugán, P. Carmona-Sáez, A. Sanchez-Pozo, Á. Rada-Iglesias, R. J. Klose, D. Landeira, Changes in PRC1 activity during interphase modulate lineage transition in pluripotent cells. Nature Communications14, 180 (2023).
2. H. G. Asenjo, A. Gallardo, L. López-Onieva, I. Tejada, J. Martorell-Marugán, P. Carmona-Sáez, D. Landeira, Polycomb regulation is coupled to cell cycle transition in pluripotent stem cells. Science advances6, eaay4768 (2020).
3. M. Espinosa-Martínez, M. Alcázar-Fabra, D. Landeira, The molecular basis of cell memory in mammals: The epigenetic cycle. Science advances10, eadl3188 (2024).
4. Y. R. Lu, X. Tian, D. A. Sinclair, The Information Theory of Aging. Nature aging3, 1486-1499 (2023).
5. G. Yang, L. Chen, G. R. Grant, G. Paschos, W. L. Song, E. S. Musiek, V. Lee, S. C. McLoughlin, T. Grosser, G. Cotsarelis, G. A. FitzGerald, Timing of expression of the core clock gene Bmal1 influences its effects on aging and survival. Science translational medicine8, 324ra316 (2016).
6. A. Gallardo, E. Belmonte-Reche, M. Marti-Marimon, J. Domingo-Reines, G. Peris, L. Lopez-Onieva, J. Xie, I. Fernandez-Rengel, P. Tristan-Ramos, N. Bellora, A. Sanchez-Pozo, A. M. Estevez, S. R. Heras, M. Marti-Renom, D. Landeira, BMAL1 represses transposable elements independently of CLOCK in pluripotent cells. Nature Communications, 16, 8250 (2025).
7. W. A. Flavahan, E. Gaskell, B. E. Bernstein, Epigenetic plasticity and the hallmarks of cancer. Science357, (2017).
8. A. Gallardo, L. López-Onieva, E. Belmonte-Reche, I. Fernández-Rengel, A. Serrano-Prados, A. Molina, A. Sánchez-Pozo, D. Landeira, EZH2 represses mesenchymal genes and upholds the epithelial state of breast carcinoma cells. Cell death & disease15, 609 (2024).
9. A. Gallardo, A. Molina, H. G. Asenjo, L. Lopez-Onieva, J. Martorell-Marugán, M. Espinosa-Martinez, C. Griñan-Lison, J. C. Alvarez-Perez, F. E. Cara, S. A. Navarro-Marchal, P. Carmona-Sáez, P. P. Medina, J. A. Marchal, S. Granados-Principal, A. Sánchez-Pozo, D. Landeira, EZH2 endorses cell plasticity to non-small cell lung cancer cells facilitating mesenchymal to epithelial transition and tumour colonization. Oncogene41, 3611-3624 (2022).
Número de expediente: IHRC22/00007
IP: Dr. David Landeira
Entidad financiadora: ISCIII
Periodo de ejecución: 01/01/2023 – 31/06/2026
Número de expediente: A-CTS-264-UGR23
IP: Dr. David Landeira
Entidad financiadora: UGR – Junta de Andalucía
Periodo de ejecución: 01/01/2024 – 31/12/2026
Número de expediente: C-CTS-274-UGR23
IP: Dr. David Landeira y Dra. Lourdes López
Entidad financiadora: UGR – Junta de Andalucía
Periodo de ejecución: 01/01/2024 – 31/12/2026
Número de expediente: PID2022-137060NB-I00
IP: Dr. David Landeira
Entidad financiadora: Agencia Estatal de investigacion
Periodo de ejecución: 01/09/2023 – 01/08/2026
Número de expediente: EMC21_00253
IP: Dra. Cristina Requena
Entidad financiadora: CUII
Periodo de ejecución: 19/09/2023 – 19/03/2028




















