
La unidad de Genómica se centra en la caracterización del genoma, la relación entre sus variantes, su expresión y su regulación. Estudiando mediante distintos abordajes la estructura de nuestro material genético, su función y evolución. Así como la integración del efecto de otros elementos genéticos y ambientales en la expresión de sus genes.
Para poder realizar estos estudios se han puesto a punto y desarrollados flujos de trabajo que nos permiten detectar SNPs, CNVs e Indels, cambios en la expresión del genoma y en la regulación de esta expresión. Dentro de este último campo hemos desarrollado técnicas que nos permiten evaluar el contenido de ARNs reguladores: lncRNAs, miRNAs, snRNAs, piRNAs. Métodos que nos permiten evaluar las modificaciones en las bases del ADN (metilación y acetilación) y la interacción del ADN con diferentes proteínas.
Gracias a la colaboración con los grupos de investigación del centro y colaboradores habituales de la Universidad de Granada y de la Consejería de Salud hemos avanzado en áreas como la farmacogenética, biopsia-líquida, caracterización de tipos celulares individualizados, determinación de microvesículas, captura de regiones en el genoma y estudios de metagenómica. Adaptando estos estudios a áreas de la salud muy diversas: oncología, enfermedades raras, cardiovasculares, inflamatorias, mentales, endocrinas; Así como en muchos casos evaluando su aplicación a la práctica clínica diaria.
La actividad de esta unidad se centra en mejorar, implementar y cooperar con la investigación en genómica de nuestro centro, así como en la Universidad de Granada y en la Consejería de Salud, sin olvidar la cooperación con nuestro entorno, otros centros de investigación y empresas. Dando preferencia a los proyectos que se encuadren en las líneas de investigación marcadas por el plan estratégico de GENYO, pero apoyando a que nuestro centro cree alianzas con otros centros.
La unidad de Genómica, dada su interacción con el resto de grupos del Parque Tecnológico de la Salud pretende realizar una investigación de excelencia, muy orientada a pacientes, con continuos procesos de mejora y buscando siempre la calidad de sus resultados. Gracias al equipamiento, los protocolos desarrollados y sobre todo a la amplia experiencia del personal científico-técnico de la unidad, asesorarán en este medio sobre las metodologías más adecuadas para cada tipo de proyecto.
Además, trataremos de apoyar la participación de los grupos de investigación de GENYO y de entidades colaboradoras en proyectos internacionales, asumiendo los paquetes de nuestra área, facilitando todo nuestro conocimiento y asesorando a otros miembros de los consorcios.
Ubicación: Planta baja
Horario: Lunes a viernes de 8:00 a 15:00
Técnicos de apoyo a la investigación: Servicio de Genómica
Clara Alcántara Domínguez
Natalia Espinar Dominguez
Juan Miguel Guerrero González
Personal asociado colaborador del Servicio de Genómica
Alicia Torres García
Técnicos de apoyo a la investigación: Servicio de Bioinformática
Raúl López Dominguez
José Manuel Gómez Silva
Contacto de la unidad de genómica:
unidad.genomica@genyo.es
Contacto de la unidad de bioinformática:
bioinfo@genyo.es
La conocida como Secuenciación de Nueva Generación (NGS) ha supuesto una revolución para el campo de la genómica, gracias al alto rendimiento con el que cuenta en comparación con tecnologías más tradicionales, ofreciendo una amplia variedad de aplicaciones para estudiar los sistemas biológicos a un nivel más complejo.
Las plataformas desarrolladas por Illumina están basadas en la secuenciación por síntesis (SBS) en la que se realiza la detección de nucleótidos marcados a medida que estos se van incorporando a la cadena en formación. En concreto, los dos sistemas con los que contamos en la Unidad de Genómica nos permiten la secuenciación de fragmentos desde 36pb hasta 600pb, y una profundidad de secuenciación que abarca desde 1 a 400 millones de lecturas por run. Como resultado de esto se obtienen datos precisos para una amplia gama de aplicaciones:
Genómica (DNA-Seq):
Transcriptómica (RNA-Seq):
Epigenómica:
Metagenómica:
El servicio básico de desarrollo de proyectos de NGS incluirá:
A través del genotipado se consigue determinar la información genética de un organismo, genotipo, poniendo de manifiesto las diferencias entre distintos genomas. En función del tipo de estudio que se desee hacer, la Unidad de Genómica dispone de distintos tipos de tecnologías para abarcar desde estudios a pequeña y mediana escala, hasta estudios que requieran tecnologías de alto rendimiento (high-throughput) que permiten los análisis de asociación de genoma completo (GWAS).
La tecnología de microarrays supone una valiosa herramienta para la detección de variantes genéticas. Además, el formato en el que se presentan permite el procesamiento de grandes volúmenes de muestras en poco tiempo. Illumina® cuenta con una amplia variedad de kits prediseñados para el genotipado de ADN humano y no humano, así como para el análisis de patrones de metilación en el ADN, ofreciendo también la posibilidad, en muchos de ellos, de customizar el ensayo.
La Unidad de Genómica cubre las posibles necesidades de todos los investigadores disponiendo de los kits prediseñados de Illumina®, entre los que podríamos destacar:
Infinium Global Screening Array-24 V3.0 Kit
Este array cuenta con aproximadamente 654.000 marcadores (con posibilidad de añadir hasta 50.000 más), proporcionando una herramienta para el genotipado a escala poblacional, la detección de variantes, estudios de farmacogenética y la investigación en medicina de precisión.
Infinium MethylationEPIC Kit
Con el Infinium MethylationEPIC array se pueden interrogar de forma cuantitativa más de 935.000 posiciones de metilación en el genoma humano a resolución de un únjico nucleótido. Permite el análisis a partir de muestras parafinadas FFPE.
Infinium CytoSNP-850K v1.2 BeadChip Kit
Este kit engloba una lista de genes basada en el contenido de la Colaboración Internacional para la Genómica Clínica (ICCG) y el Consorcio de Genómica del Cáncer (CGC), de manera que, con hasta 850.000 marcadores, proporciona una visión general y completa de genes de reconocida relevancia citogenética tanto en aplicaciones convencionales como de cáncer.
Para más información sobre las posibilidades que esta casa comercial ofrece visite https://www.illumina.com/index-d.html.
El servicio de análisis de genotipado y metilación con microarrays de Illumina® incluye:
La PCR digital supone un nuevo enfoque en la detección de ADN que proporciona una cuantificación absoluta y la detección de dianas de forma más eficiente y precisa que la PCR cuantitativa, todo ello sin la necesidad de usar estándares o patrones de referencia. Desde su aparición ha sido fundamental para detectar variantes raras, proteínas de fusión y para el trabajo en biopsia líquida.
En la Unidad de Genómica contamos con el equipo QuantStudio Absolute Q Digital PCR System de Applied Biosystems™, para llevar a cabo las reacciones de PCR digital, el cual genera datos absolutos de cuantificación con una elevada precisión y sensibilidad.
Entre las aplicaciones que esta tecnología permite encontraríamos:
La PCR en tiempo real es una técnica ampliamente empleada para numerosas aplicaciones tanto cualitativas como cuantitativas. Para llevar a cabo es tipo de ensayos contamos con hasta 4 plataformas diferentes: ABI Prism 7900HT, ABI Prism 7500, QuantStudio™ 6 Flex Real-Time PCR System y QuantStudio™ 12K Flex System, de Applied Biosystems™.
Los servicios ofertados dentro de esta categoría englobarían:
Basada también en la PCR a tiempo real, la tecnología OpenArray™ permite llevar a cabo análisis de expresión génica, identificación de genotipos de SNPs, análisis de microRNAs o ensayos de farmacogenómica a través de ensayos TaqMan™ prediseñados o nuevos que pueden ser seleccionados para adaptarse a los requerimientos de su experimento.
En función del análisis a realizar, existen hasta 11 posibilidades de configuración de experimentos prediseñados adaptados a nuestro sistema QuantStudio™ 12K Flex System, de Applied Biosystems™.
Para más información sobre el diseño de experimentos, visite la web de ThermoFisher: https://www.thermofisher.com/es/es/home/life-science/pcr/real-time-pcr/real-time-openarray/open-array-panel.html.
A través del sistema Bioanalyzer 2100 y TapeStation 4200 de Agilent podemos determinar la calidad de los ácidos nucleicos, tanto ADN como ARN, de forma automatizada mediante un proceso de electroforesis de alta resolución.
Este procedimiento se ha convertido en punto imprescindible en muchos de los flujos de trabajo de un laboratorio de genómica, destacando sobre todo como control de calidad de la cantidad, integridad y pureza del ADN y ARN en los procesos de generación de librerías para NGS, requiriendo volúmenes muy pequeños de muestra.
Aplicaciones del análisis de ácidos nucleicos en Bioanalyzer 2100 y TapeStation 4200:
Sonicación
Los equipos de sonicación se han convertido en un instrumental esencial en cualquier laboratorio dedicado a la investigación en genómica. Con el E220 Focused-Ultrasonicator, de Covaris®, conseguimos una alta precisión en el control de todo el proceso gracias a la tecnología AFA que consigue dirigir la onda al centro del pocillo.
Entre las aplicaciones que nos permite esta tecnología encontramos:
PCR de punto final
En la Unidad de Genómica contamos con varios equipos para desarrollar PCR convencional: Veriti™ Dx Thermal Cycler y GeneAmp® PCR 9700, de Applied Biosystem™.
Estos equipos se encuentran repartidos en dos áreas físicamente separadas para cubrir todo el rango de aplicaciones en los que son necesarios. Así, un área de pre-amplificación (libre de amplicones), en la que se utilizarán en procesos como generación de librerías para NGS, períodos de incubación en protocolos de purificación de muestras, proceso de conversión bisulfito para estudios de patrones metilación, etc. Los ubicados en el área de post-amplificación, serán usados para la amplificación de regiones de interés del genoma en estudio.
Análisis espectrofotométrico y fluorométrico de ácidos nucleicos
Casi la totalidad de las aplicaciones que se desarrollan en un laboratorio de genómica requieren de pautas previas que den información sobre la calidad y la concentración de las muestras.
Contamos con diferente equipamiento para obtener esta información:
Para más información acerca de los servicios ofertados, así como de las condiciones para el envío de muestras puede ver aquí cómo contactar con nosotros, o escribiendo directamente a unidad.genomica@genyo.es.
Durante la última década las denominadas tecnologías ómicas han supuesto una profunda revolución en la investigación biomédica. Métodos como la secuenciación masiva, los DNA microarrays o las técnicas de proteómica y metabolómica están permitiendo a los investigadores estudiar los mecanismos celulares desde una perspectiva global y han demostrado ser de una enorme utilidad para caracterizar los procesos moleculares asociados a diferentes patologías. Estas nuevas tecnologías están generando enormes cantidades de datos por lo que para su almacenamiento, procesado e interpretación se hace necesario el uso de avanzadas técnicas estadísticas y de minería de datos, así como el manejo de recursos computacionales de altas prestaciones que permitan llevar a cabo una gestión eficiente de los mismos. Es este contexto el que ha hecho que la Bioinformática se haya convertido en una disciplina esencial en todos los centros de investigación genómica.
Las principales líneas de trabajo del Servicio de Bioinformática se centran en:
- Soporte en el análisis e interpretación de datos ómicos a través de técnicas bioinformáticas de vanguardia mediante protocolos referentes en su ámbito:
- Arrays de metilación
- Secuenciación de ARN
- Secuenciación de small ARN
- Secuenciación de ADN – búsqueda de variantes
- Secuenciación de ADN – metagenómica
- GWAS
- Experimentos de célula única
- Consultoría en el diseño experimental
- Almacenamiento de datos
- Elaboración y consulta acerca de la generación de figuras para publicaciones científicas
Para más información acerca de los servicios de bioinformática ofertados, puede contactar con nosotros directamente a bioinfo@genyo.es
La Unidad de Genómica del centro GENYO centra su labor en el marco de la genómica funcional y genotipado aplicados a la salud. Son muchas las técnicas que pueden desarrollar gracias al conjunto de científicos y técnicos especialistas, dedicados al estudio integral del ADN, su expresión y la regulación de ésta. Para desarrollar su labor cuenta con plataformas y equipos para realizar todas las técnicas de una forma automatizada, consiguiendo la mayor cantidad de información en el menor tiempo posible y minimizando al máximo el número de errores. Además de contar con acuerdos y colaboraciones para poder llevar a cabo todo tipo de proyecto, sea cual sea la tecnología necesaria y la magnitud del mismo. Algunas de las plataformas disponibles son las siguientes:
Secuenciación de nueva generación
NextSeq 500 (Illumina®), es una plataforma de nueva generación para secuenciación masiva de ADN. Permite la secuenciación de genomas completos, exomas, paneles dirigidos y transcriptomas, entre otros, con muy alto rendimiento, basado en la reconocida química “Sequencing By Synthesis” (SBS).
El sistema NextSeq 500 es muy versátil. Permite la secuenciación de media a alta escala, lo que hace que pueda adaptarse a multitud de proyectos distintos sin comprometer su rendimiento y su eficiencia óptimos. Permite la realización de estudios de perfil de la expresión génica, mRNA-Seq, análisis de pequeños ARNs, estudios de epigenómica, secuenciación dirigida de genes, secuenciación de exomas y grandes paneles, además de la secuenciación de genomas completos.

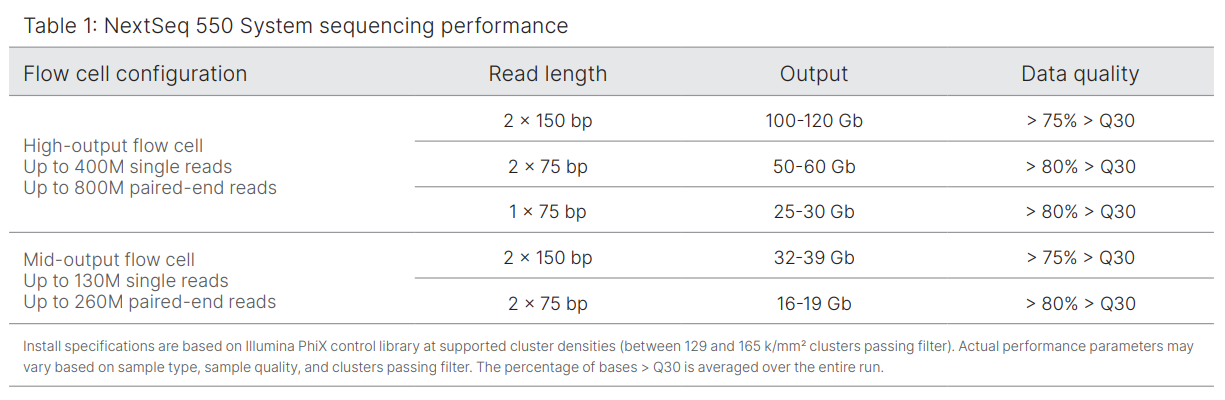
MiSeq (Illumina®), es otra plataforma de NGS basada también en la tecnología SBS, capaz de ofrecer datos de alta precisión y un rendimiento sólido para una amplia gama de aplicaciones: secuenciación de genes dirigidos, genomas pequeños y amplicones, metagenómica 16S, etc.
Es capaz de realizar lecturas paired-end de hasta 15 Gb por carrera, proporcionando datos de secuencias de más de 600 bases por lectura. Una plataforma compacta todo en uno que incorpora además análisis de datos y acceso a Sequence Hub de BaseSpace™ (la plataforma de análisis genómico de Illumina).

Scanners de Microarrays de alto rendimiento
La plataforma iScan (Illumina®) es un escáner de alta resolución de microarrays basado en la tecnología “Bead-Based microarray”.
Cuenta con micromatrices cubiertas por cientos de miles de microesferas que portan copias de oligos específicos. Estos actúan como secuencias de captura para una amplia gama de aplicaciones para el análisis de ADN.
El sistema iScan va acompañado de una amplia variedad de ensayos y kits para genotipado de genoma completo o específico, perfiles de metilación y análisis citogenético, disponibles desde la web de Illumina: https://emea.illumina.com/systems/array-scanners/iscan/products-services.html

Sistemas de cuantificación de expresión génica y genotipado por PCR a tiempo real
Dos equipos QuantStudio™ 6 Flex Real-Time PCR System, Applied Biosystems™ con bloques para 96 y 384 pocillos. Los sistemas QuantStudio cuenta con la tecnología OptiFlex, la cual permite el multiplexing avanzado para la cuantificación de múltiples genes dentro del mismo experimento. Este sistema permite realizar en la misma secuencia ensayos basados tanto en TaqMan como en SYBR, facilitando una mayor flexibilidad en el diseño experimental.
Sistema QuantStudio™ 12K Flex System with OpenArray™. Permite analizar de 1 a 12.000 reacciones en una sola carrera, y hasta 110.000 reacciones en un solo día, cuando se combina con el sistema AccuFill QuantStudio 12K Flex OpenArray. Es una tecnología que abarata el coste y reduce el tiempo de manipulación.

Dos plataformas 7900HT de Applied Biosystems™, con bloques para 96 y 384 pocillos y un bloque para tarjetas microfluídicas. Sistemas de PCR a tiempo real con capacidad para el análisis de 1-30 posiciones en 384 muestras. Además, un sistema de PCR a tiempo real 7500 de Applied Biosystems™, plataforma calibrada para una amplia gama de fluoróforos disponibles: FAM™⁄SYBR™ Green I, VIC™⁄JOE™, TAMRA™⁄CY3, ROX™⁄Texas Red y CY5.


PCR digital
Applied Biosystems™ lanza en 2022 su nuevo sistema para PCR digital, el QuantStudio Absolute Q Digital PCR System. Esta nueva plataforma se basa en la tecnología de placa de matriz microfluídica (MAP) que permite concentrar la mayor parte del flujo de trabajo en un único instrumento (compartimentación, ciclo térmico y adquisición de datos), minimizando los pasos prácticos y maximizando la uniformidad de los ensayos.

Resto del equipamiento disponible
Dado el carácter multidisciplinar de GENYO la Unida de Genómica del centro ha tenido la oportunidad de colaborar, desde su apertura, con universidades, centros de investigación y empresas en numerosos proyectos de investigación tanto nacionales como internacionales. Esta colaboración consiste en dar servicio a los investigadores proporcionándoles asesoramiento técnico y/o en realizar el estudio que requiera cada proyecto: estudios de asociación de Genoma completo (GWAS), genotipado, CNVs, epigenética, perfiles de expresión génica, interacción DNA/proteína, diversidad microbiana, etc.
Entre las colaboraciones que tiene la Unidad de Genómica con diferentes entidades podríamos destacar las siguientes:
Centros I+D
Empresas
Empresas extranjeras
A lo largo de los años la Unidad de Genómica ha participado en numerosos proyectos que le han permitido aumentar su cartera de servicios y especializarse en técnicas, crear grandes alianzas con multitud de grupos de investigación de distintas áreas de conocimiento, y en definitiva, crecer como Unidad ofreciendo un servicio de calidad, personalizado y de confianza.
Algunos de los proyectos más relevantes en los que ha colaborado y colabora la Unidad de Genómica son:
 3TR
3TR
Identification of the molecular mechanisms of non-response to treatments, relapses and remission in autoimmune, inflammatory, and allergic conditions
The 3TR is the largest immunology project funded by the Innovative Medicine Initiative 2 (IMI 2) to date and it will focus on autoimmune, inflammatory and allergic and chronic diseases.
3TR will bring together interdisciplinary experts from 69 academic and industrial partner institutions covering 15 European countries. The Scientific Coordinator of 3TR is Dr. Marta Alarcón Riquelme, Head of Medical Genomics at the GENYO centre at the Fundación Pública Andaluza Progreso y Salud.
Many patients with autoimmune, inflammatory and allergic diseases do not respond well or at all to current treatments. A major challenge for researchers is to understand which patients are most likely to respond to which treatments. The aim of 3TR is to shed new light on the factors that determine whether or not a patient is likely to respond to a given treatment. 3TR will focus on seven diseases: systemic lupus erythematosus (SLE), rheumatoid arthritis, multiple sclerosis, ulcerative colitis, Crohn’s disease, asthma, and chronic obstructive pulmonary disease (COPD). Despite their diverse symptoms, these diseases have some molecular patterns in common, suggesting that patients with different diseases may share some markers that predict the likelihood of treatment response and disease progression.
The project will analyse data and samples from 50 000 patients who took part in 50 clinical trials. They will also carry out a new clinical study that will take samples (of diseased tissue, blood, stool and other fluids) from patients before, during and after treatment. The project will add all of its data to a centralised data management platform that will make it easier to analyse using state-of-the-art methods. The samples collected and the knowledge base will be maintained beyond the end of the project to allow further research. The project will ultimately make it easier to provide the right treatment to the right patient, thereby increasing the proportion of patients taking a treatment that works for them.
 PRECISESADS
PRECISESADS
Molecular Reclassification to Find Clinically Useful Biomarkers for Systemic Autoimmune Diseases
PRECISESADS’ consortium represents a range of partner organisations of high scientific reputation in the field, and with experience in undertaking large human cohort studies, 2 SMEs, and 5 major pharmaceutical companies. The coordinator of the consortium is Dr. Chris Chamberlain, VP Head of Experimental Medicine and Diagnostics at UCB. The managing entity is led by Dr. Marta E. Alarcón-Riquelme, Head of the Department of Medical Genomics at GENYO (Genomic and Oncological Research centre), administered by the FPS.
The goal of this EU project is the use of the power of OMICs, and bioinformatics to identify new classifications for diseases known to share common pathophysiological mechanisms (systemic lupus erythematosus (SLE), systemic sclerosis (SSc), Sjögren’s syndrome (Sjs), rheumatoid arthritis (RA), primary antiphospholipid syndrome (PAPS) and mixed connective tissue disease (MCTD), jointly as systemic autoimmune diseases (SADs)). Such knowledge has not been applied to individual patients, depriving them from potential benefits in terms of the use of new therapeutic agents that are being developed for one disease but cannot be applied to another due to current clinical classifications.
Results will be widely shared to deliver a new molecular taxonomy of systemic autoimmune diseases that can be directly accessed by physicians, patients, regulators and drug developers to help define, refine and discover better treatments for SADs. In order to achieve this, they are informing punctually and on a regular basis on the project implementation and results, and involve stakeholders from Europe in its activities in order to broaden the projects’ impact and increase opportunities for cooperation.
http://www.precisesads.eu/about-the-project/
 U-PGx
U-PGx
UBIQUITOUS PHARMACOGENOMICS
The GENYO Genomics Unit collaborates closely with the Clinical Management Unit of Hospital Pharmacy at the Hospital Universitario Clínico San Cecilio (Granada) (Dr. Cristina Dávila Fajardo) in the European proyect UPGx (UBIQUITOUS PHARMACOGENOMICS).
The U-PGx consortium will address major challenges and obstacles for implementation of pharmacogenomic (PGx) testing in patient care, taking into account the diversity of healthcare systems and citizens across Europe. Specifically, U-PGx will investigate if the emerging approach of pre-emptive
genotyping of an entire panel of important PGx markers is cost-effective and results in a better outcome for patients. With the pre-emptive PGx testing approach data on multiple important pharmacogenes are collected prospectively and embedded into the patients’ electronic record. Typically, it alerts prescribers and pharmacists through electronic clinical decision support systems when a drug is ordered or dispensed for a patient with an at-risk genotype. The new model of personalised medicine through pre-emptive PGxtesting will be conducted at a large scale in seven existing European health care environments (The Netherlands, Spain, UK, Italy, Austria, Greece, Slovenia).
http://upgx.eu/
 PISMA-ep
PISMA-ep
Estudio epidemiológico de la salud mental en Andalucía
La Unidad de Genómica de GENYO colabora estrechamente en la parte técnica del proyecto con dos de las investigadoras del estudio PISMA-ep: Dra. Blanca Gutiérrez del Centro de Investigación Biomédica en Red de Salud Mental (CIBERSAM), Departamento de Psiquiatría, Facultad de Medicina, Universidad de Granada y Dra. Margarita Rivera perteneciente a la Unidad de Salud Mental, Complejo Hospitalario Universitario de Granada, Instituto Biosanitario de Granada.
El estudio PISMA-ep se ha llevado a cabo en las 8 provincias de Andalucía, la mayor comunidad autónoma española con casi 9 millones de habitantes censados, lo que representa casi una quinta parte del total de la población española. El estudio PISMA-ep es el primer estudio epidemiológico centrado en los trastornos mentales y sus factores asociados, y realizado en una muestra representativa de toda la población andaluza. Este estudio fue una iniciativa del Plan Integral de Salud Mental de Andalucía (PISMA), oficina del Servicio Andaluz de Salud con especial interés en medir la comorbilidad psiquiátrica y sus correlatos en Andalucía con el fin de obtener datos para una planificación futura. Los objetivos principales del estudio PISMA-ep fueron: 1) estimar las prevalencias transversales y de vida de trastornos mentales frecuentes; 2) explorar los factores sociales, psicológicos y genéticos asociados con estos trastornos mentales, y 3) estudiar una cohorte transversal grande que pudiera constituir la base para un futuro seguimiento prospectivo o para estudios prospectivos.
 InmunoSIGHT
InmunoSIGHT
Caracterización inmunológica y molecular en biopsia líquida de pacientes de cáncer de pulmón para inmunoterapia
La Unidad de Genómica de GENYO colabora estrechamente en la parte técnica del proyecto con la empresa Atrys Health, cuya sede en Granada está ubicada dentro del Centro.
El objetivo principal del presente proyecto es el desarrollo de una herramienta que permita caracterizar desde un punto de vista inmunológico y molecular a los pacientes de cáncer de pulmón no microcítico con el fin de encontrar una firma molecular que permita la estratificación de pacientes frente a la inmunoterapia, integrando datos clínicos, moleculares e inmunológicos propios del tumor y que identifique los largos supervivientes con estos tratamientos, actualmente imposible de reconocer de antemano con ningún parámetro conocido.
Para ello, se reclutará una cohorte de 150 pacientes diagnosticados con cáncer de pulmón no microcítico, estadio IV a fin de investigar su estado inmunitario y la relación con la supervivencia a la inmunoterapia, prestando especial atención a aquellos pacientes considerados largos supervivientes (> 2 años de supervivencia global).
http://www.atryshealth.com/es/inmunosight_15121
 GENEIDA
GENEIDA
Genetics, Environmental Exposures and Infant Development in Andalusia
La Unidad de Genómica de GENYO participa junto a Escuela Andaluza de Salud Pública, ibs.GRANADA (Dra. Marina Lacasaña) y Hospital de Poniente en el proyecto GENEIDA.
La exposición prenatal a diferentes factores adversos, tanto ambientales, como nutricionales y genéticos pueden contribuir a un mayor riesgo de padecer complicaciones en el periodo neonatal, y a un incremento en la mortalidad perinatal. Además, la exposición intrauterina a dichos agentes se ha asociado a un mayor riesgo de enfermedades durante la infancia y la vida adulta.
El análisis del efecto de las exposiciones tempranas sobre el crecimiento y desarrollo fetal e infantil se realizará en el marco de la cohorte de nacimiento GENEIDA (Genetics, Environmental Exposures and Infant Development in Andalusia), constituida en el Poniente Almeriense desde abril de 2014, cuyo objetivo es la evaluación del efecto de las exposiciones tempranas adversas (compuestos tóxicos ambientales, estrés psicológico y social, enfermedades maternas, malnutrición fetal, así como factores genéticos y epigenéticos) en el desarrollo las enfermedades a lo largo de la vida.
 GESTAFIT
GESTAFIT
GESTAtion and FITness. Efectos de un programa de ejercicio físico supervisado durante el embarazo sobre la longitud de los telómeros y marcadores de expresión génica relacionados con la adiposidad en la madre y el neonato
La Unidad de Genómica de GENYO colabora estrechamente en la parte técnica del proyecto GESTAFIT con la investigadora la Dra. Virginia Aparicio del departamento de Fisiología de la Universidad de Granada.
Dicho proyecto es pionero a nivel internacional estudiando los efectos de un programa de ejercicio concurrente y de la dieta sobre diversos marcadores moleculares, genéticos y epigenéticos, de biogénesis mitocondrial, perfil inflamatorio, glucémico y lipídico, etc., todo ello en múltiples tejidos (suero de madre y neonato, placenta, leche, etc.). En él, también se valoran otras esferas relacionadas con la salud, tales como el dolor, la calidad del sueño, la ecografía muscular, la salud positiva, la salud mental o la calidad de vida durante el embarazo. Estos estudios van a resaltar la importancia de la actividad física durante el embarazo para un mejor estado de salud materno-fetal y la prevención de complicaciones perinatales.
La continuación de dicho proyecto se centrará en el estudio de la descendencia (estudio GESTAFITOS).
Se sabe que durante el periodo intrauterino se produce la denominada “programación fetal”, en la que el entrenamiento a lo largo de la gestación pudiera haber tenido un efecto modulador positivo. Por tanto, el ejercicio podría haber encendido o apagado interruptores (expresión de ciertos polimorfismos genéticos) que podrían favorecer una descendencia más sana.
 ONCOFERT
ONCOFERT
Identificación de variantes génicas de recuperación de la espermiogénesis postquimioterapia tras cáncer en varones
La Clínica IVI Sevilla, la Universidad de Jaén y la Unidad de Genómica de GENYO trabajaron conjuntamente para desarrollar una nueva herramienta diagnóstica que permitiera predecir qué pacientes iban a tener mayor o menor probabilidad de recuperar su función reproductiva, y en su caso, aconsejarles o no los servicios de preservación de gametos antes de someterse al tratamiento quimioterápico.
El objetivo del proyecto era buscar e identificar variantes génicas tipo SNPs encontradas en el exoma mediante su genotipado en muestras de sangre de pacientes sometidos a quimioterapia hacía más de 2 años que habían recuperado la fertilidad frente a los que no la habían recuperado, pudiendo dar lugar a potenciales marcadores moleculares que permitieran pronosticar la recuperación de la fertilidad de varones con cáncer testicular o Linfoma No Hodgkin sometidos a quimioterapia.
 TRADIONP
TRADIONP
Prototipo de sistema experto para el tratamiento y diagnóstico oncológico personalizado
La medicina personalizada se basa en identificar cohortes de pacientes analizando sus homologías moleculares, y que sean susceptibles de ser tratadas bajo los mismos criterios.
En este estudio retrospectivo, se analizaron muestras de pacientes con cáncer de mama, cáncer colorrectal y cáncer de pulmón que habían sido sometidos a dos regímenes terapéuticos: Terapia con agentes químicos y Terapias Biológicas.
Para la consecución de este estudio se construyó un Consorcio de Excelencia formado por las empresas Indra, Althia y Lorgen y la colaboración especial de GENYO, en el que la sinergia de conocimientos y tecnologías de cada miembro permitió evaluar el uso y la eficacia en Oncología de modalidades diagnósticas y terapéuticas avanzadas en el marco sanitario de la Comunidad Autónoma de Andalucía.
El objetivo de este proyecto era la creación de un nuevo estándar de gestión integral del paciente oncológico para guiar la terapia multimodal (cirugía, radioterapia y quimioterapia), de forma personalizada y eficaz. La consecución de este objetivo se abordó técnicamente mediante la creación de una base de datos multimodal que alberga información a partir de los datos de enfermos oncológicos (datos clínicos, datos histopatológicos, perfiles moleculares (genotipado y epigenética) y datos de la imagen médica).
 Intranet
Intranet



































